Unsere Arbeitsgruppe beschäftigt sich mit der Entwicklung, Charakterisierung und Anwendung von molekularen Werkzeugen für die hochpräzise Fluoreszenzmikroskopie, insbesondere im Bereich der Super-Resolution Imaging Verfahren. Unser Ziel ist es, biologische Strukturen in ihrer funktionellen, zellulären Umgebung mit sub-10-nm Auflösung sichtbar zu machen – quantitativ, dynamisch und molekular exakt.
Biologisch-kompatible Präzisionslabelingstrategien
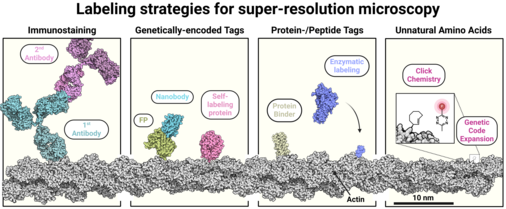
Ein zentrales Problem moderner Mikroskopie ist der Trade-off zwischen optischer Präzision und biologischer Relevanz: Die besten Farbstoffe sind meist synthetisch, aber schwierig zu platzieren; genetisch enkodierbare Labels sind präzise, aber optisch limitiert. Wir entwickeln und kombinieren Strategien, um diese Lücke zu schließen:
-
Genetische Code-Expansion zur Inkorporation nicht-kanonischer Aminosäuren in definierte Positionen funktionaler Proteine.
-
Bioorthogonale Klick-Chemie Reaktionen, insbesondere Tetrazin-Ligationen, die schnelle, fluorogene, zytokompatible Markierung ermöglichen.
-
Peptid- und Enzym-Tags mit minimalem sterischem Einfluss, darunter HALO- und SNAP-tag-Varianten, die wir gezielt modifizieren
-
Fluorophore mit optimierten photophysikalischen Eigenschaften – hohe Photostabilität, kontrollierbares Photoswitching und Fluorogenität
Durch diese Kombination können wir funktionserhaltendes, site-spezifisches Labeling in lebenden Zellen etablieren – mit minimalem Linkage Error, hoher Signalqualität und definierter Stöchiometrie.
Molekulare Auflösung & Quantitative Super-Resolution Imaging
Die Abbildung molekularer Strukturen mit Auflösung jenseits der optischen Beugungsgrenze verlangt neben geeigneter Markierung auch methodische Innovation auf der Detektionsseite. Wir entwickeln und nutzen:
-
Photoswitching-basierte Verfahren wie dSTORM und PAINT mit 1–5 nm Lokalisationspräzision.
-
Fingerprinting-Ansätze zur Klassifizierung einzelner Moleküle auf Basis ihrer Schaltkinetik
-
Molekulare Nanolineale, z. B. auf Basis von PCNA oder DNA-Origamis, zur Kalibration und Validierung von Mikroskopen auf Sub-10-nm-Niveau.
-
Quantitative Analysepipelines zur Bildsegmentierung, Lokalisationsstatistik und Vergleich funktioneller Proteinzustände.
Wir arbeiten daran, Imaging nicht nur „scharfer“, sondern auch quantifizierbar und standardisierbar zu machen – bis auf die molekulare Ebene.
Translationale Anwendungen & molekulare Funktion
Unsere Verfahren erlauben nicht nur strukturelle Kartierung, sondern auch funktionelle Untersuchung dynamischer Protein-Protein-Interaktionen, Rezeptoraktivierungen und Signaltransduktion in zellulären Systemen. Aktuelle Anwendungsbeispiele:
-
Rezeptordynamik & Aktivierung bei GPCRs
-
Visualisierung von Pseudoviren und Infektionsmechanismen
-
Analyse mitochondrialer Funktion und neuronaler Kommunikation in primären Zellen und Gewebeschnitten
-
Labeling schwieriger Zielproteine wie AMPA-, GluK-, NMDA- und GABA-Rezeptoren in neuronalen Kulturen
Unsere Expertise wird zunehmend auch für kollaborative Projekte im Bereich der translationalen Forschung und Wirkstoffentwicklung genutzt – etwa zur Visualisierung von Wirkstoffbindung, zur Identifikation therapeutisch relevanter Konformationszustände oder zur bildbasierten Charakterisierung von Pathomechanismen bei neuropsychiatrischen Erkrankungen.

Perspektiven
Unsere Forschung integriert Chemie, Pharmazie, Optik, Zellbiologie und Strukturbiologie zu einem interdisziplinären Programm mit dem Ziel, die molekulare Architektur und Dynamik biologischer Systeme sichtbar zu machen – so präzise wie nötig, so funktional wie möglich.
Wir bauen derzeit an einer offenen Plattform für molekulare Bildgebung, in der Fluorophore, Tags, Mikroskopieprotokolle und Referenzstandards modular kombiniert werden können. Diese Plattform soll zukünftig auch kollaborativ genutzt und erweitert werden – als Beitrag zu einer standardisierten, molekular validierten Bioimaging-Technologie für die Lebenswissenschaften.
